La técnica CRISPR de edición genética está de moda. Parece ser la piedra filosofal que permitirá desarrollar múltiples e importantes aplicaciones en campos diversos como la medicina, biotecnología, agricultura o ganadería pero, quizás, lo más revolucionario sea su uso para almacenar información en células vivas. Dos investigadores españoles, Patricia Rada Llano y Juan Antonio Calles tienen mucho que decir en esta línea de investigación que quiere convertir el ADN en un disco duro y nos aproxima a alguno de los excéntricos futuros mostrados en la serie Black Mirror. Patricia Rada es Doctora en Bioquímica, Biología Molecular y Biomedicina y Juan Antonio Calles es doctor en Informática y CEO de la compañía especializada en ciberseguridad ZeroLynx. Hemos hablado con Juan Antonio para que nos explique algo más sobre su revolucionario trabajo.
– En primer lugar, me gustaría saber qué trabajos previos les han servido de inspiración, a usted y a la doctora Patricia Rada, para el desarrollo de esta técnica.
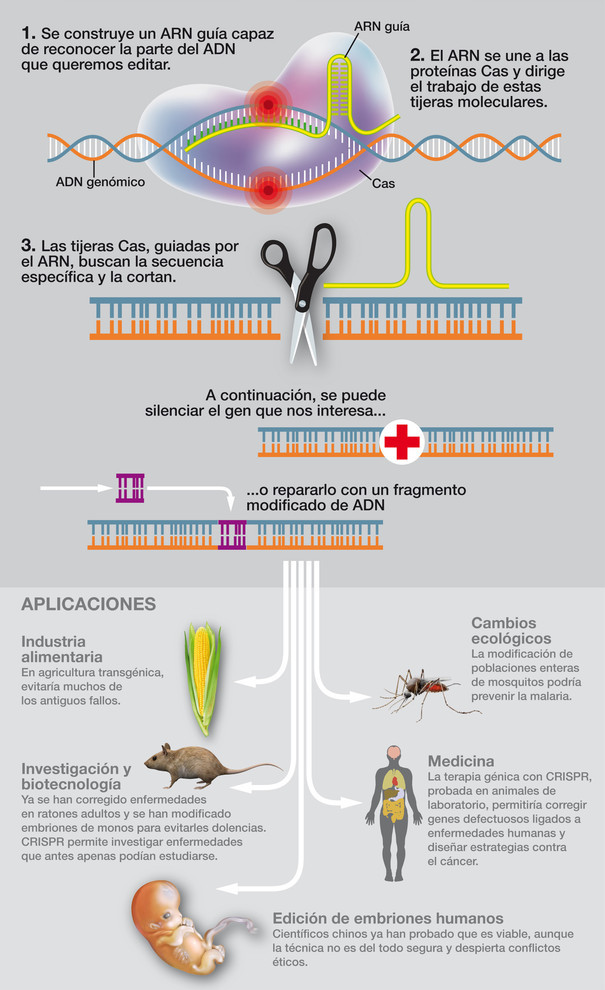
En el verano del año pasado se publicó un gran trabajo en una de las revistas científicas más importantes del mundo dentro del campo de la Biomedicina, la revista Nature, que explicaba cómo el grupo de investigación del Dr. G.M. Church había sido capaz de codificar varias imágenes y fotogramas en el ADN bacteriano mediante la técnica CRISPR, descubierta por el español Francis Mójica en el año 2005.
Los sitios CRISPR son secuencias de ADN viral integradas en el cromosoma bacteriano. Estas secuencias contienen fragmentos de ADN de virus que han infectado a las bacterias en una primera infección. Estos fragmentos son reconocidos por la bacteria durante una segunda infección, de manera que activa un mecanismo de destrucción de estas partículas virales. Por tanto, el sistema CRISPR/Cas se trata de un sistema inmune procariótico que confiere resistencia a agentes externos como plásmidos y fagos? y provee una forma de inmunidad adquirida.
Así, estas secuencias juegan un papel clave en los sistemas de defensa bacterianos, y forman la base de una tecnología conocida como CRISPR / Cas9, que hoy en día se aplica como sistema de edición de genes en múltiples organismos.
Cuando estuvimos analizando este trabajo, vimos que desde el punto de vista bioquímico era un paso de gigante que iba a ser la base de muchos avances futuros, pero nos dimos cuenta que desde el punto de vista informático podría ser mejorado a nivel de compactación y de automatización.
via GIPHY
GIF introducido en el genoma de bacterias por investigadores de la Universidad de Harvard.
– ¿Nos puede explicar, de la forma más sencilla que sea posible, en qué consiste?
En el trabajo que hemos presentado en RootedCon 2018, hemos mostrado el software Bacter10, una aplicación que hemos desarrollado para generar automáticamente las secuencias de bases de ADN -denominadas técnicamente protospacers, y compuestas por A, T, G, C, las letras del código genético- necesarias para introducir en el ADN bacteriano los datos que nos pueda interesar partiendo de un archivo cualquiera (una imagen, un video o un fichero de texto, por ejemplo).
En unas décadas, con la mejora de la tecnología y mayor investigación, se podría almacenar todos los datos de internet en 90 gramos de ADN humano.
– ¿Qué ventajas ofrece respecto a las técnicas actuales de almacenamiento de datos?
Hoy en día, el límite de almacenamiento de datos en bacterias con estas técnicas es aún teórico, debido a las restricciones técnicas del sistema. Sin embargo, cabe esperar que, en unas décadas, gracias a la mejora de la tecnología y con mayor investigación, podamos almacenar hasta 455 exabytes por gramo de ADN.
Sólo para hacer una estimación, el ADN de una sola célula humana que contiene 3.3 x 10^9 bases, es decir, 3.3 mil millones de bases, pesa tan sólo 3.6 picogramos. Es decir, cada humano tenemos alrededor de 200 gramos de ADN en nuestro interior. De acuerdo con la IDC (International Data Corporation), se calcula que el volumen de datos mundial de Internet alcanzará los 40 Zetta Bytes, ZB (40.000.000.000 Tera Bytes) en 2020. Por tanto, en base a estos cálculos, podríamos estimar que estos 40 ZB podrían almacenarse en ~90 gramos de ADN.

– ¿En qué punto se encuentra ahora su desarrollo?
Partiendo de los trabajos anteriormente presentados, hemos dado continuidad al mismo desarrollando un software que permite codificar y decodificar archivos informáticos partiendo directamente de ficheros extraídos de un ordenador, como pueda ser una imagen, por ejemplo. El siguiente paso sería poder generar estas secuencias e introducirlas en estos organismos. Para ello nos encontramos en la actualidad buscando patrocinio, ya que el coste de esta fase del trabajo es demasiado alto como para asumirlo con nuestros medios actuales.
– ¿Qué coste supone ahora esta técnica de almacenamiento y cuándo podrá ser rentable?
Actualmente, el coste sería elevado puesto que antes de llevar al mercado este proyecto necesitaríamos hacer varios experimentos en el laboratorio para garantizar que podemos trasladar las secuencias generadas por ordenador a un organismo vivo con una exactitud en torno al 100%, puesto que no debemos olvidar que el objetivo final es tener copias exactas de la información que nos interesa conservar. El proceso conllevaría en primera instancia la realización y posterior validación de que cada paso que realizamos es correcto, y así continuar con el siguiente.
Una vez asegurados que los procedimientos están optimizados, lo siguiente sería la implantación del sistema de una manera más automatizada que nos permitiera rebajar los costes para, en el menor plazo de tiempo posible, poder hacer de este proyecto un producto viable para el almacenamiento de nuestra valiosa información.
– ¿Cómo preservar el material genético de microorganismos o bacterias vivas en el que se almacena la información? ¿Qué condiciones, físicas y de seguridad, son necesarias?
Según la cepa bacteriana, tendríamos que ajustar las condiciones. En el caso del trabajo al que hacemos referencia, se trata de la cepa de Escherichia coli BL21, cuyo nivel de bioseguridad es bajo, con lo que únicamente necesitaría de unas condiciones asequibles de crecimiento, como temperatura constante a 37 ºC y con un medio enriquecido de nutrientes.
Una de las principales ventajas es que la población bacteriana tiene una alta tasa de replicación con lo que podríamos ser capaces en un período corto de tiempo de tener muchas copias de nuestra información y lo que es más importante, almacenarla en diferentes sitios para las copias de seguridad que deseemos. Asimismo, muchos organismos tienen maquinaria propia de corrección de errores durante la replicación, siendo por tanto un sistema robusto de almacenamiento de información.

– ¿Cuál es el desarrollo del Biohacking en nuestro país? ¿Existe una red de investigadores consolidada con aportaciones relevantes?
En las últimas décadas la informática se había puesto al servicio de la bioquímica y la biomedicina, mediante el desarrollo de hardware y software para facilitar el trabajo diario de los investigadores. Termocicladores, sistemas de revelado, etc. son algunos de los ejemplos de cómo la tecnología y la informática en general han ayudado de forma muy positiva a la bioquímica y a la medicina. Pero en estos últimos años estamos viendo la aproximación inversa, en la que las ciencias anteriores se están poniendo al servicio de la informática para integrar de una manera más natural la vida y la tecnología, haciendo más cercanos aún si cabe algunos de los futuros excéntricos presentados en series de televisión como Black Mirror. España es una cuna de grandísimos investigadores en ambos campos, recordemos entre otras aportaciones de relevancia que el CRISP surgió gracias al descubrimiento de un español, el Dr. Francis Mójica. Pero está claro que esta unión es compleja y requiere un profundo conocimiento de ambas disciplinas, que en raras ocasiones suelen confluir en una única persona, por lo que las colaboraciones son fundamentales, y es algo que debe motivarse a nivel gubernamental, y no únicamente a nivel privado, con becas y ayudas a los investigadores y a los laboratorios.
Los avances deben ir encaminados a lograr un almacenamiento estable en organismos vivos, en abaratar su coste y que los procesos de codificación y decodificación sean asumibles para más grupos de investigación.
– ¿Ha mostrado ya interés alguna empresa o institución por su trabajo? ¿Se plantean generar negocio a partir de su hallazgo? Algunas empresas se nos han acercado para interesarse por la tecnología que hemos desarrollado, aún en periodo de pruebas, pero por el momento no tenemos nada cerrado. Nos encontramos ahora mismo en busca de empresas que quieran patrocinar nuestra investigación o colaborar en ella con sus recursos.
– Es interesante la transversalidad de su proyecto que une la biología y la informática, ¿cree que se potencia suficientemente en la universidad esta “contaminación” entre disciplinas distintas para aportar soluciones complejas a realidades cada vez más complejas? ¿Cómo fue su encuentro con Patricia Rada?
A excepción de algunas titulaciones que están surgiendo en los últimos años, y de algunas asignaturas impartidas en los grados de química, biología y bioquímica sobre el uso de lenguajes de programación tipo Python para desarrollar algoritmos, se puede decir que esa “contaminación” positiva aún es muy limitada. Sin embargo, en los últimos años en las carreras de ciencia se están implantando cada vez más asignaturas de bioinformática y nanotecnología, lo que amplía el espectro considerablemente. Pensamos que es necesario que ambos mundos colaboren y se entiendan, para que proyectos como bacter10 puedan evolucionar. En nuestro caso, Patricia y yo nos conocíamos desde hacía varios años, y habíamos pensado con anterioridad en colaborar en algún proyecto juntos, pero no nos lo planteamos realmente hasta que nuestro amigo Josep Albors, Head of Awareness and Research de ESET España, nos propuso este reto y nos lanzamos a la piscina.
– ¿Cuáles serán los siguientes pasos esa unión de la biología con la informática? Háganos de futurólogo.
Nos encontramos en un punto demasiado temprano como para valorar una fecha estimada para poder desarrollar discos duros biológicos basados en bacterias o en cualquier otro tipo de organismo vivo. Por el momento los avances deben ir encaminados a lograr un almacenamiento estable en organismos vivos, y en mejorar el procedimiento para abaratar su coste y que los procesos de codificación y decodificación sean asumibles para más grupos de investigación. De esta manera podrá aumentar la investigación y quizás hablemos en algunas décadas de discos duros “vivos”.



hola: soy Israel Rubio de México, seria bueno que el comité de ética quitara sus restricciones en humanos , para poder hacer estudios para que las personas no enfermen, se regeneren en todo, cabello, órganos, y no tenga que envejecer, Dios nos dio toda la capacidad para ser mejores cada día, la ciencia no tiene limites,! si vamos a tener equivocaciones! pero siempre con la conciencia de que se esta haciendo el bien y Dios no va juzgar a las buenas acciones por que es en bien de la humanidad, todo tiene su madurez, la nuestra yo soy técnico programador en sistemas informáticos, si en verdad estudiamos para lograr algo por favor permitan que se estudie y se investigue usted Dra. Rada comparta con todos los que queremos ética sin restricciones, en la naturaleza existen errores y no por eso ya dejamos de existir de ser, gracias por su apreciable tiempo mucho éxito